-
摘要:目的 鉴定并比较不同测序方案中多发性骨髓瘤(multiple myeloma,MM)潜在的驱动基因,为探讨MM的发生机制提供研究基础。方法 选择两套数据,一套数据为2016年5月至2020年11月苏州大学附属第二医院84例MM患者的基因组靶向突变测序数据,另一套数据从癌症基因组图谱(The Cancer Genome Atlas, TCGA)数据库下载的205例MM全基因组测序数据。在R4.1.0环境下,利用驱动基因识别算法对MM的潜在驱动基因进行挖掘,分析并比较两套数据筛选出的驱动基因及具体突变信息。结果 NRAS、KRAS、TP53、IDH1为两套数据共同识别出的驱动基因。两套数据中突变基因在NOTCH、RTK-RAS、Cell-Cycle、TP53、MYC及WNT通路均有富集。显著突变的NRAS、KRAS、TP53和IDH1在两套数据中发生突变的位点无显著性差异。另外,通过靶向突变测序方法,鉴定出MM新的驱动基因,CBL、BCOR和DNMT3A。结论 通过本研究获得了MM可能的驱动基因,有助于从基因分子层面揭示MM发生发展机制。Abstract:Objective The potential driver mutation genes of multiple myeloma (MM) in different sequencing protocols were identified to provide a research basis for exploring the pathogenesis of this disease.Methods This study used two cohorts. A cohort of 84 patients with MM from The Second Affiliated Hospital of Soochow University was subjected to genome-targeted mutation sequencing. The other cohort (205 patients with MM) was selected from The Cancer Genome Atlas (TCGA) using whole-genome sequencing. The potential driver genes of MM were mined using the driver gene identification algorithm in the R4.1.0 environment. Subsequently, the driver genes and mutation spectrum were analyzed.Results NRAS, KRAS, TP53, and IDH1 were identified in the two datasets. Furthermore, the mutated genes in both datasets were enriched in the NOTCH, RTK-RAS, Cell-Cycle, TP53, MYC, and WNT pathways. The recurrent mutation sites in NRAS, KRAS, TP53, and IDH1 showed no significant difference between the two datasets. In addition, new driver genes of MM were identified via targeted mutation sequencing, these are CBL, BCOR, and DNMT3A.Conclusions In this study, we obtained the potential driver genes of MM, which are helpful for revealing the pathogenesis of this disease.
-
Keywords:
- multiple myeloma (MM) /
- mutation /
- driver gene /
- target sequencing
-
多发性骨髓瘤(multiple myeloma,MM)是一种恶性增殖的浆细胞肿瘤,其特征是克隆性浆细胞无限增殖,在恶性血液肿瘤发病率中占第二位[1]。近年来,随着蛋白酶体抑制剂、免疫调节剂和CD38单克隆抗体等药物以及自体干细胞移植越来越多的应用于临床,MM患者的预后得到极大的改善[2-3]。然而,仍有部分患者并不能从目前的治疗中获益,无法达到有效缓解[4],提示MM患者的精准分层及个体化治疗是目前亟待解决的问题。
肿瘤是由基因突变驱动的疾病,随着高通量测序技术和癌症基因组学的快速发展,研究人员能够快速分析转录组和基因组的全貌,揭示数目庞大的体细胞突变并找到驱动肿瘤发生发展的关键基因[5]。这些关键基因当以极小的概率发生突变时,将导致恶性克隆扩增。关键基因发生突变将赋予癌细胞生长优势和生存能力,进而驱动肿瘤的发生发展,因此被称为驱动基因[6]。MM患者的特征之一是由于染色体不稳定性引起的拷贝数和结构变化导致的细胞遗传学异常,这是影响 MM患者预后的一大重要因素[7]。由此识别潜在的驱动基因将为骨髓瘤患者的个性化靶向治疗提供可能性[8],对于降低MM的发病率和改善患者的预后将具有重要意义。
在本研究中,首先从84例患者的含有118个基因集合的二代测序数据出发,建立1个标准合理的流程来识别潜藏在MM患者样本中的驱动基因,并联合来自癌症基因组图谱MM患者的全基因组测序数据,分析并比较两套数据筛选出的驱动基因及具体突变信息。这些发现将为MM提供新的重要的基因组改变特征,为后续深入研究MM的发病机制提供生物信息学依据。
1. 材料与方法
1.1 临床资料
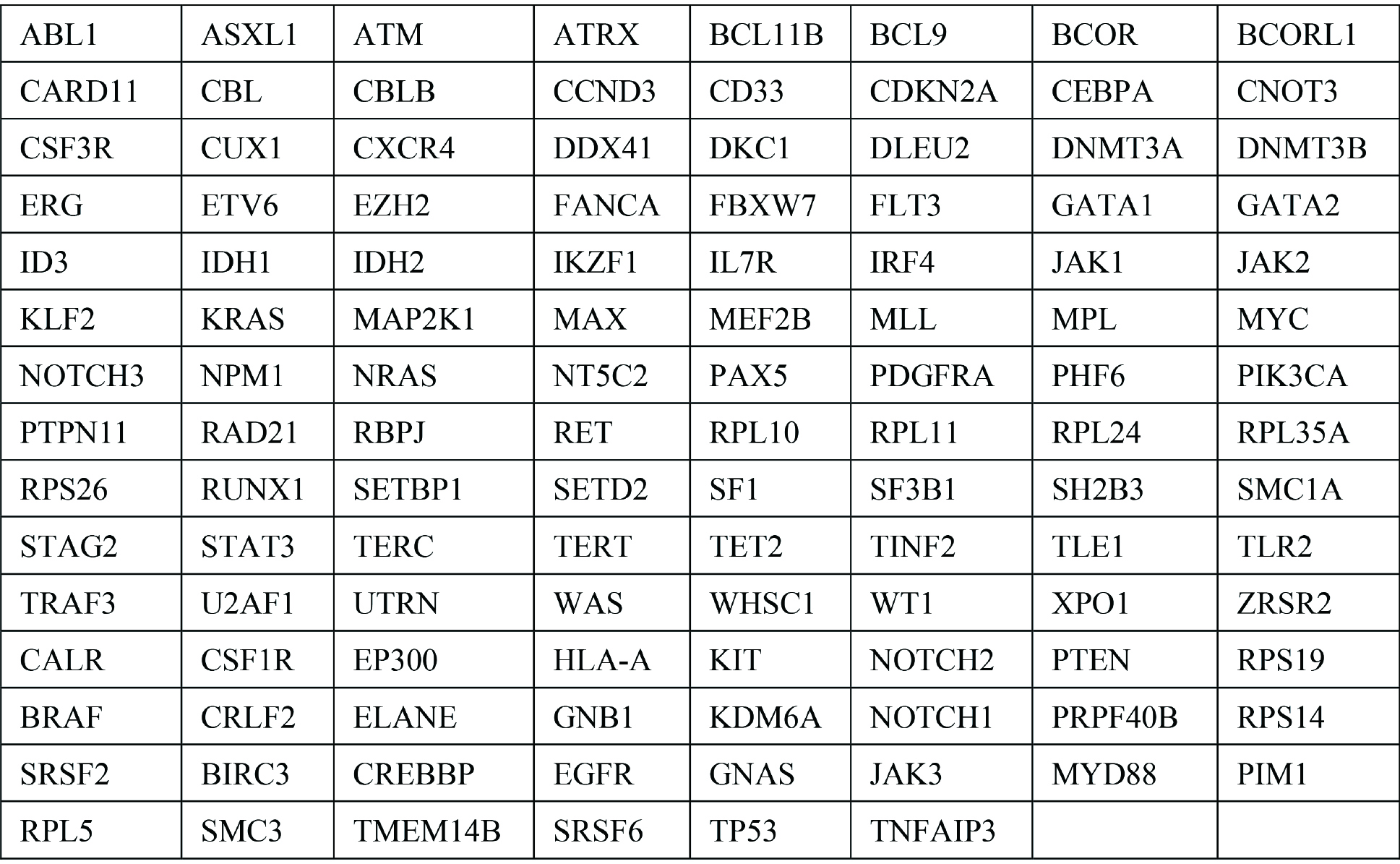
回顾性分析2016年5月至2020年11月在苏州大学附属第二医院诊断并接受治疗的84例MM患者初诊时的骨髓单核细胞二代测序结果,采用与MM发生发展密切相关的118个基因为靶向测序集合。此外,利用TCGA数据库 (https://portal.gdc.cancer.gov/) 下载了205例MM患者的全基因组测序数据[9],见图1。本研究通过了苏州大学附属第二医院伦理学审查委员会的批准。
1.2 方法
OncodriveCLUST为一款基于突变聚类的驱动基因识别软件,主要针对功能获得性突变,这些突变通常聚集在蛋白质的特定区域,具有形成突变簇的偏好性,通过对这些突变进行分析来寻找潜在的驱动基因[10]。使用OncodriveCLUST软件对MM患者的突变数据进行分析,统计蛋白质上每个位置的功能获得性突变的频率分布并进行聚类,利用每个基因上的簇,对基因进行打分。最后根据P<0.05, 错误发现率(false discovery rate,FDR)<0.1筛选可能的驱动基因。
SomInaClust是根据突变模式从全外显子组或基因组突变数据中识别候选驱动基因[11]。基本假设是由于选择压力,驱动基因的特征为1)聚类突变;2)肿瘤样本中大量的失活(蛋白质截断)突变。前者是原癌基因 (proto-oncogene,OG) 的主要模式,后者是抑癌基因(tumor suppressor genes,TSG)的主要模式。随后使用Benjamini & Hochberg 方法对OG和TSG的P值进行多次测试校正。根据校正后的P值(qOG和qTSG),将驱动基因定义为qDG<0.05的基因。
MutSigCV主要考虑到肿瘤的异质性,首先根据突变信息建立在肿瘤形成过程中起作用的背景突变模型,根据模型判断每个基因的突变是否比偶然突变频率更高[12]。使用算法软件MutSigCV对MM的突变基因进行分析,算出MM多发性点突变基因发生频率的P值和q值,根据q<0.05筛选出驱动基因。
1.3 统计学分析
采用R语言4.1.0版本对数据进行统计学分析。P值均基于双向假设检验,以P<0.05为差异具有统计学意义。
2. 结果
2.1 驱动基因筛选
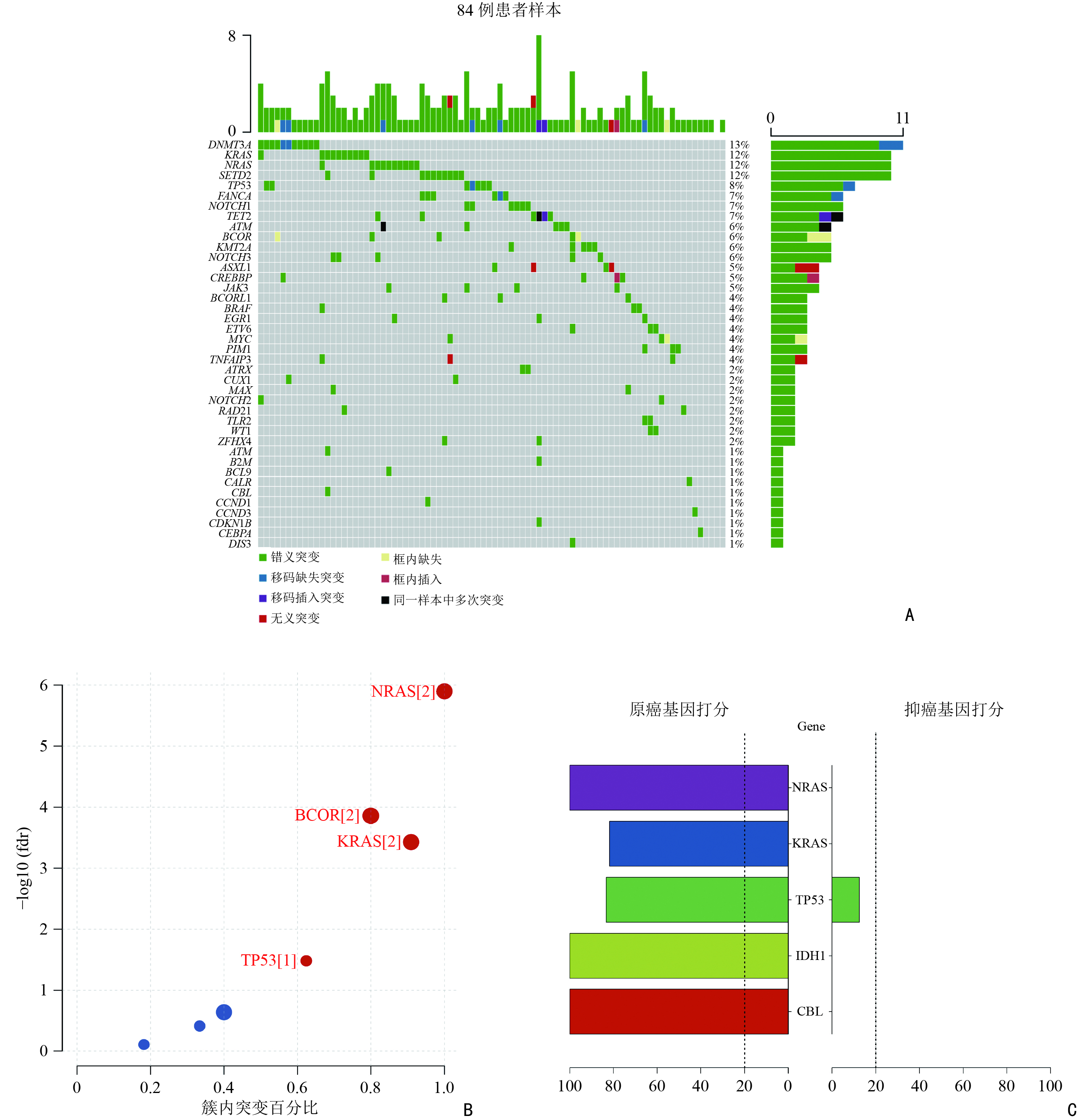
首先本研究使用R中maftools程序包[13]可视化展示84例MM患者的突变信息,如图2A所示,按突变频率排序展示前40个基因。本研究采用3种方法筛选驱动基因。首先,使用OncodriveCLUST软件识别驱动基因[10],主要针对功能获得性突变进行分析,这些突变通常聚集在蛋白质的特点区域,可能是肿瘤细胞生长优势和肿瘤细胞克隆进化过程中正向选择的信号,通过对这些突变进行分析,来预测潜在的驱动基因。见图2B,NRAS、BCOR、KRAS、TP53被识别为MM中的驱动基因。其次,使用癌症基因检测软件SomlnaCLUST[11]识别驱动基因,这是一种基于驱动基因在肿瘤样本中的突变模式准确识别驱动基因,并进一步将其分类为癌基因或肿瘤抑制基因的方法。结果显示,NRAS、KRAS、IDH1、CBL为驱动基因,并被划分为原癌基因(图2C);与其他研究结果相一致的是TP53具有较高的原癌基因分数[14-15]。最后,本研究应用基于频率的MutSigCV进行突变负荷分析寻找MM的驱动基因[12]。驱动基因信息如表1所示,以q<0.05为阈值,NRAS、KRAS、DNMT3A、TP53被识别为驱动基因。
表 1 Mutsigcv识别驱动基因基因 N_非沉默的 N_沉默的 N_非编码 N_非沉默的 N_沉默的 N_非编码 χ2 P q NRAS 113967 29403 0 11 0 0 0 9.16E-11 1.73E-06 KRAS 138834 33453 0 11 0 0 0 4.07E-10 3.84E-06 DNMT3A 563193 154143 0 11 0 0 0 3.21E-07 2.02E-03 TP53 250209 69093 0 8 0 0 0 1.27E-06 6.01E-03 由于本研究所提供的84个MM组织样本的二代测序集合仅包含与MM发生发展密切相关的118个基因,虽节省人力、物力、财力及时间,但识别出的驱动基因有限。因此为了验证此种方法的可靠性,本研究从癌症基因图谱(TCGA,https://cancergenome.nih.gov/)获取了205例MM患者样本的全基因组测序数据[9],进行驱动基因识别并比较两套数据的基因突变信息。
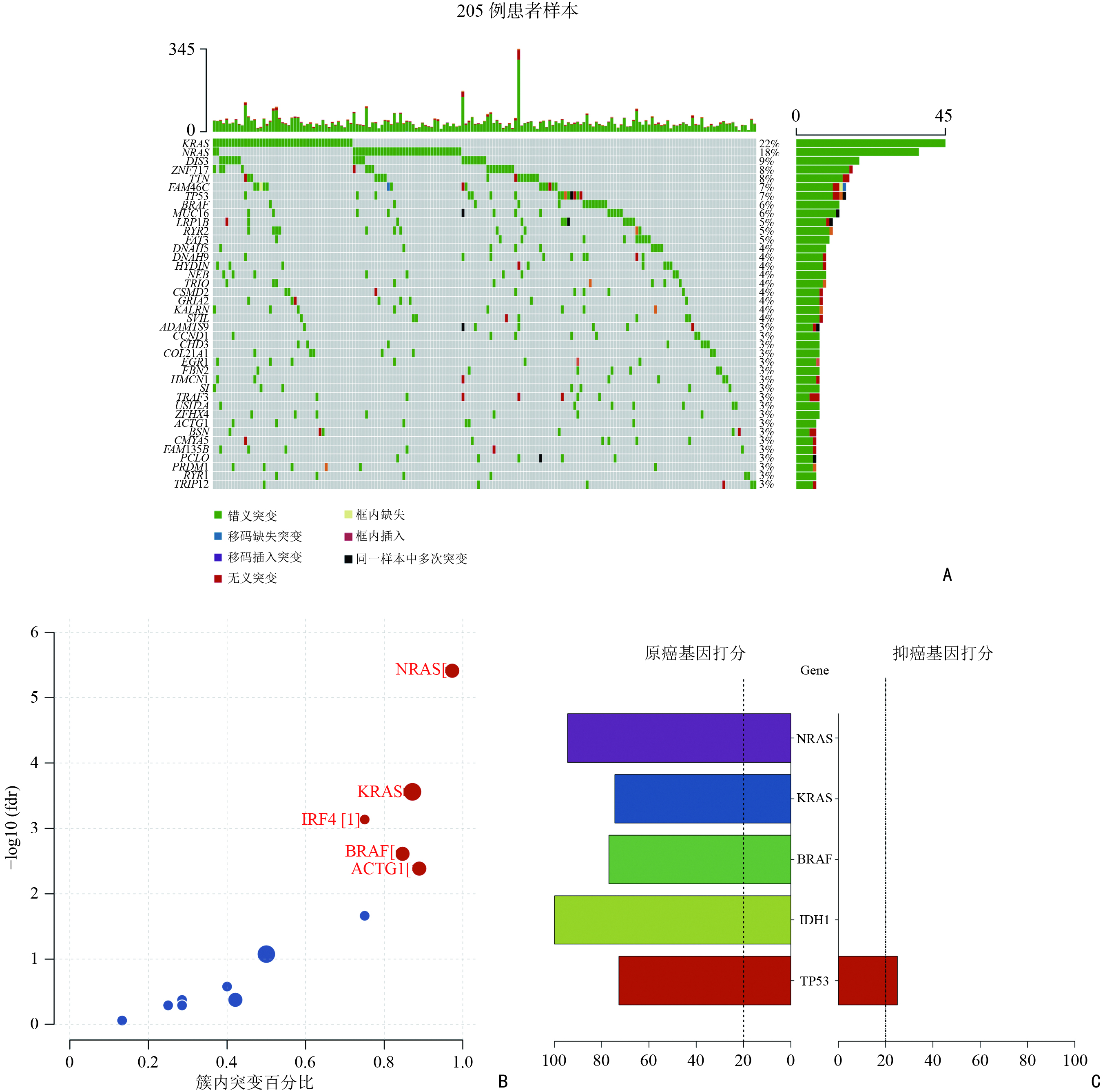
如图2所示,首先使用maftools将205例MM患者的突变信息进行可视化展示,结果显示KRAS、NRAS等基因的突变频率较高(图3A),这与靶向突变测序结果一致。接下来使用OncodriveCLUST、SomlnaCLUST、MutSigCV软件分别寻找驱动基因。OncodriveCLUST识别出NRAS、KRAS、IRF4、BRAF和ACTG1为驱动基因(图3B)。SomlnaCLUST识别出NRAS、KRAS、BRAF、IDH1为原癌基因,TP53同样具有较高的原癌基因分数(图3C)。 KRAS、NRAS、TP53、DIS3、ZNF717、BRAF、TRAF3被MutSigCV识别为驱动基因(表2)。分析结果可知,NRAS、KRAS、TP53、IDH1为两套数据共同识别出的驱动基因。
表 2 Mutsigcv识别驱动基因基因 N_非沉默的 N_沉默的 N_非编码 N_非沉默的 N_沉默的 N_非编码 χ2 P q KRAS 351370 84665 0 47 0 0 5 0 0 NRAS 288435 74415 0 37 0 0 5 0 0 TP53 633245 174865 0 16 0 0 3 4.29E-13 2.70E-09 DIS3 1477435 385400 0 19 1 0 4 7.65E-09 3.61E-05 ZNF717 1366940 330460 0 18 2 0 10 4.06E-07 1.53E-03 BRAF 1137340 320825 0 13 0 0 4 1.08E-06 3.38E-03 TRAF3 858130 216275 0 9 0 0 3 1.35E-05 3.64E-02 2.2 两套数据基因突变信息的比较
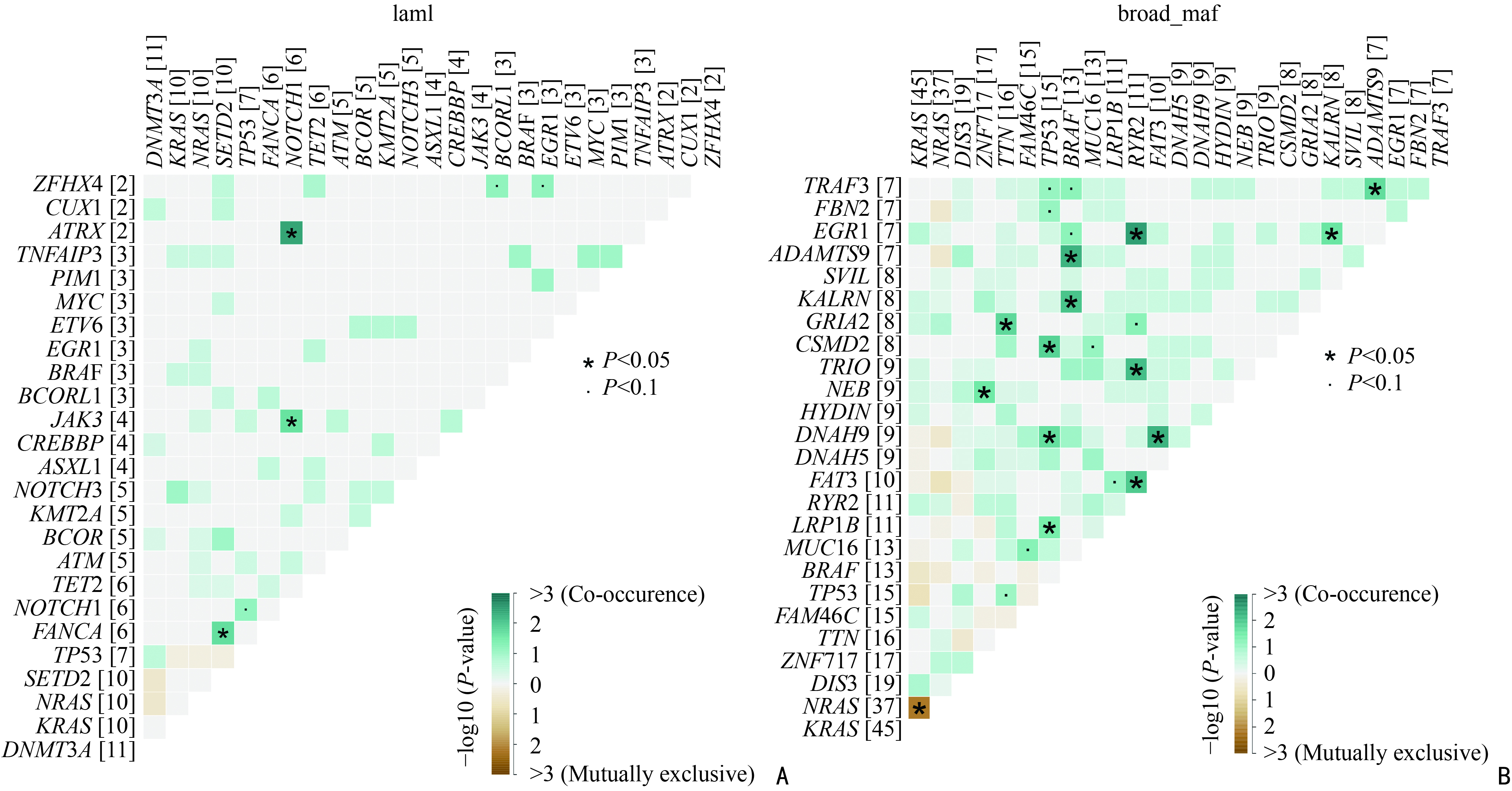
为更好地比较两套MM患者的基因突变情况,本研究采用coOncoplot函数接受两个对象并将它们并排绘制成条形图。结果如图3所示,两套数据均显示,KRAS、NRAS和TP53的突变频率高。本院84例MM患者的靶向测序结果中,DNMT3A突变频率最高(图4A)。随后将突变基因进行致癌信号通路的富集,结果显示两套MM患者的突变基因在NOTCH、RTK-RAS、Cell-Cycle、TP53、MYC及WNT通路均有富集(图4B)。许多致癌基因具有比其他基因座更频繁突变的优先位点,这些位点被认为是突变热点,棒棒糖图可以用于显示突变热点以及其他突变位点。利用Lollipopchart将单个基因的突变位点及其所在蛋白结构域进行可视化展示,并比较两套患者基因突变数据中显著突变的NRAS、KRAS、TP53和IDH1。结果如图4C所示,两套数据中高频突变的基因其发生突变的位点一致。
![]() 图 4 两套数据基因突变信息的比较laml为84例MM患者的突变数据,broad_maf为205例MM患者的突变数据。A:条形图展示突变频率较高的8个基因在两套数据中的突变比例,左边laml为84例患者的突变数据,右边broad_maf为205例患者的突变数据,下方颜色条代表不同的基因突变类型。无义突变、错义突变、框内缺失、剪接位点突变和同一样本中多次突变;B:展示两套MM患者的突变基因分别富集到的通路。左边laml为84例患者的突变数据,右边broad_maf为205例患者的突变数据;C:棒棒糖图直观显示基因在蛋白质结构上的突变位点。上方laml为84例患者的突变数据,下方broad_maf为205例患者的突变数据
图 4 两套数据基因突变信息的比较laml为84例MM患者的突变数据,broad_maf为205例MM患者的突变数据。A:条形图展示突变频率较高的8个基因在两套数据中的突变比例,左边laml为84例患者的突变数据,右边broad_maf为205例患者的突变数据,下方颜色条代表不同的基因突变类型。无义突变、错义突变、框内缺失、剪接位点突变和同一样本中多次突变;B:展示两套MM患者的突变基因分别富集到的通路。左边laml为84例患者的突变数据,右边broad_maf为205例患者的突变数据;C:棒棒糖图直观显示基因在蛋白质结构上的突变位点。上方laml为84例患者的突变数据,下方broad_maf为205例患者的突变数据2.3 基因突变之间共同发生和相互排斥的模式
为观察两套数据中基因突变的模式,本研究采用somaticInterations绘制基因间相互排斥或共发生事件的情况。如图5A所示,在84例MM患者靶向测序数据中,以P<0.05为阈值,本研究发现突变基因中NOTCH1与ATRX、JAK3;FANCA与SETD2存在显著共发生模式。而在205例MM患者突变基因中存在共发生与相互排斥的基因明显较多(图5B)。这可能与样本的统计量及基因的统计量有关。
2.4 驱动基因的鉴定
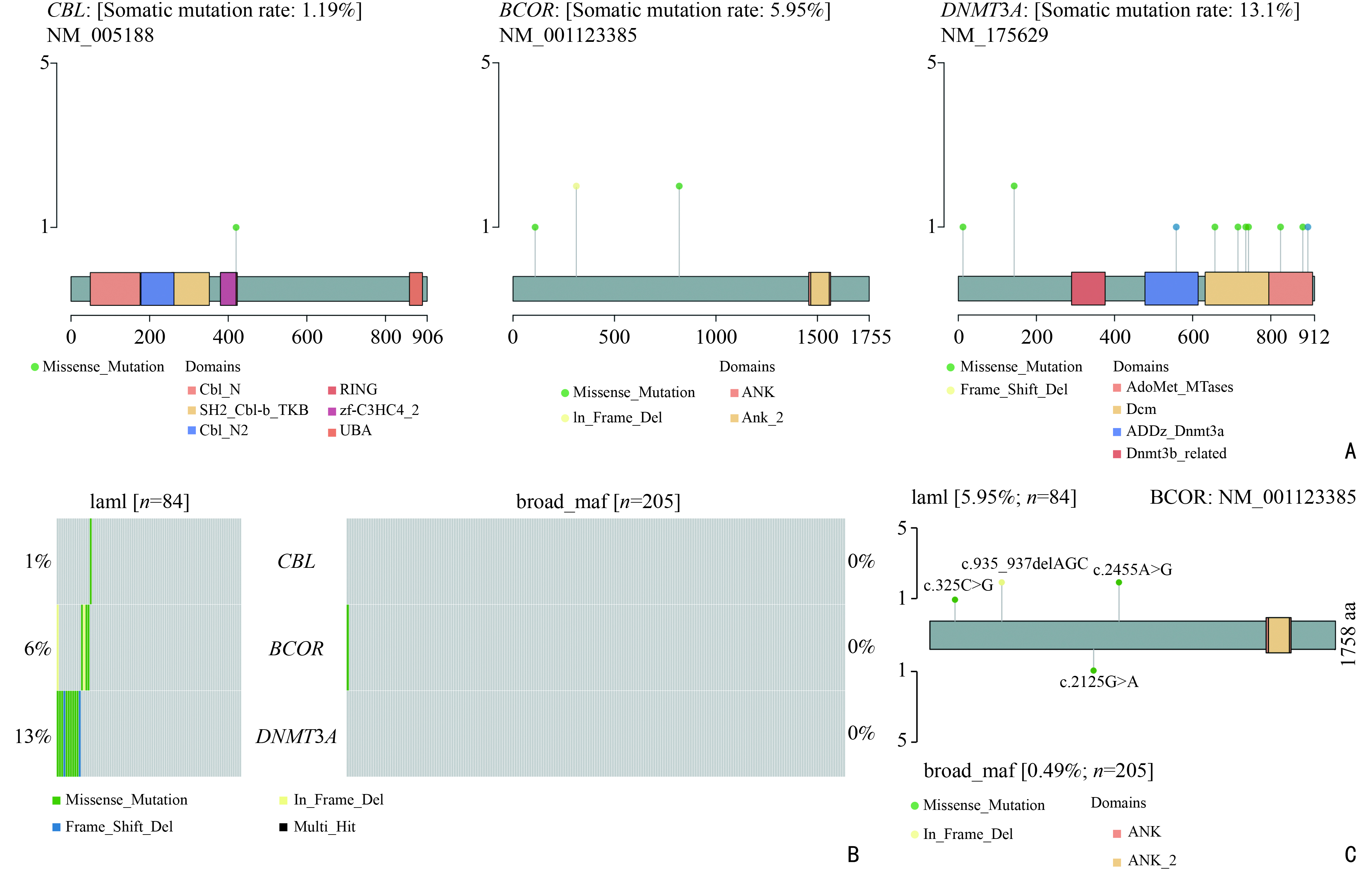
通过比较来自本院的84例和来自TCGA数据库的205例MM患者突变数据的驱动基因分析结果,研究发现CBL、BCOR和DNMT3A是新鉴定出的驱动基因,并将其突变位点绘制如下(图6A)。已有研究报道DNA甲基转移酶DNMT3A(DNA methyltransferase 3 alpha,DNMT3A)通过调控DAB2IP介导的MEK/ERK激活促进结直肠癌进展[16]、且DNMT3A在急性髓系白血病(acute myeloid leukemia,AML)患者中具有较高突变频率,并且与不良预后密切相关[17]。此外,原癌基因CBL(Cbl proto-oncogene,CBL)的突变通过增加与LYN和PIK3R1的相互作用驱动致癌信号通路PI3K/AKT[18]。BCL6辅抑制因子(BCL6 corepressor)BCOR最近也被证明其突变在成熟T细胞淋巴瘤中具有高复发性和致癌性[19]。此外,本研究针对CBL、BCOR和DNMT3A 3个新鉴定的驱动基因在两套数据中的差异表现进一步分析。结果显示在205例MM患者中,CBL、DNMT3A无突变,BCOR中仅1例突变(图6B)。随后将BCOR在两套患者基因突变数据中的突变位点通过棒棒糖图可视化展示并比较,结果如图6C所示,两套患者基因突变数据中BCOR的突变位点无重合,由此推测本选取的差异和样本统计量有限。
3. 讨论
MM是一种分子水平异质性很大的恶性肿瘤,在精准医疗模式下,分子分型的差异在很大程度上决定了患者的个体化治疗方案[20-21]。目前,对于MM的分子机制的研究越来越多,但由于其调控网络的复杂性,需要对MM发生发展过程中的关键驱动因子做更深入的挖掘,寻找特异性更高的标志物,以期为骨髓瘤患者提供更好的治疗方案。
癌症驱动因素的识别是解释癌症发生机制和实现精准医疗的关键挑战。根据单个突变位点或整个基因来识别癌症驱动因素的方法有很多,但各具优缺点。MutSigCV基于基因突变的频率,通过识别基因与背景突变的关系来定义驱动基因。背景突变模型用于量化乘客突变的积累,过高或过低的背景突变率的估计会导致结果不准确。同义突变常被用作固定的背景模型。然而,研究表明背景突变并不是均匀分布的。因此,准确估计背景突变率仍然具有挑战性。OncodriveCLUST,一种识别蛋白质序列中具有显著突变聚类倾向的基因的方法。该方法通过编码沉默突变构建背景模型,将背景率阈值以上多个突变的位置识别为潜在有意义的簇。该方法识别出具有功能获得性突变的癌基因,但在预测以功能丧失性突变为特征的肿瘤抑制基因方面不足。然而,以上两种方法通常不能检测不太频繁的突变基因,也不能区分原癌基因或肿瘤抑制基因。SomInaClust是一种基于驱动基因在肿瘤样本中的突变模式来准确识别驱动基因,并进一步将其分类为癌基因或肿瘤抑制基因的方法。该方法使用参考突变数据:1)确定突变热点,即蛋白编码区(coding sequences,CDS )位置在研究样本中包含比偶然预期更多的突变;2)计算基因背景突变率。SomInaClust是对其他候选驱动基因识别方法的补充。
从比较的结果看出,MutSigCV、OncodriveCLUST、SomInaClust可以互相发现一些其他检测方法忽略的基因。因此,在检测肿瘤的驱动基因时,除去样本量是影响算法性能的稳定性外,建议使用基于不同原理的软件综合检测。
在本研究中,首先以回顾性分析的本院初诊的84例MM患者的118个基因的靶向二代测序数据为参考数据,使用基于突变聚类、突变模式及突变频率的算法识别潜在的驱动基因。而后分别将上述寻找驱动基因的算法应用于TCGA数据中。通过分析并比较两套体细胞突变数据筛选出的驱动基因,结果显示在84例使用靶向测序集合的体细胞突变数据中驱动基因:NRAS、BCOR、KRAS、TP53、IDH1、CBL、DNMT3A;在205例使用全基因组测序的TCGA体细胞突变数据中驱动基因:IRF4、ACTG1、BRAF、IDH1、KRAS、NRAS、TP53、DIS3、ZNF717、TRAF3。本研究发现NRAS、KRAS、TP53、IDH1为两套数据共同识别出的驱动基因。随后,通过比较两套数据中具体的突变信息,本研究观察到KRAS、NRAS和TP53的突变频率显著较高。通路富集分析发现两套MM患者的突变基因在NOTCH、RTK-RAS、Cell-Cycle、TP53、MYC及WNT通路均有富集。比较两套患者基因突变数据中高频突变的NRAS、KRAS、TP53和IDH1,发现在两套MM数据中发生突变的位点一致。此外,通过比较来自本院的84例和来自TCGA数据库的205例MM患者突变数据的驱动基因分析结果,本研究找出CBL、BCOR和DNMT3A为新鉴定的驱动基因。
综上所述,此研究通过多数据整合的方法,鉴定了MM的潜在驱动基因,为MM提供新的重要的基因组改变特征。本研究所鉴定出的驱动基因可能为今后进一步通过科研实验手段探索MM发病机制提供新的线索。
-
图 4 两套数据基因突变信息的比较
laml为84例MM患者的突变数据,broad_maf为205例MM患者的突变数据。A:条形图展示突变频率较高的8个基因在两套数据中的突变比例,左边laml为84例患者的突变数据,右边broad_maf为205例患者的突变数据,下方颜色条代表不同的基因突变类型。无义突变、错义突变、框内缺失、剪接位点突变和同一样本中多次突变;B:展示两套MM患者的突变基因分别富集到的通路。左边laml为84例患者的突变数据,右边broad_maf为205例患者的突变数据;C:棒棒糖图直观显示基因在蛋白质结构上的突变位点。上方laml为84例患者的突变数据,下方broad_maf为205例患者的突变数据
表 1 Mutsigcv识别驱动基因
基因 N_非沉默的 N_沉默的 N_非编码 N_非沉默的 N_沉默的 N_非编码 χ2 P q NRAS 113967 29403 0 11 0 0 0 9.16E-11 1.73E-06 KRAS 138834 33453 0 11 0 0 0 4.07E-10 3.84E-06 DNMT3A 563193 154143 0 11 0 0 0 3.21E-07 2.02E-03 TP53 250209 69093 0 8 0 0 0 1.27E-06 6.01E-03 表 2 Mutsigcv识别驱动基因
基因 N_非沉默的 N_沉默的 N_非编码 N_非沉默的 N_沉默的 N_非编码 χ2 P q KRAS 351370 84665 0 47 0 0 5 0 0 NRAS 288435 74415 0 37 0 0 5 0 0 TP53 633245 174865 0 16 0 0 3 4.29E-13 2.70E-09 DIS3 1477435 385400 0 19 1 0 4 7.65E-09 3.61E-05 ZNF717 1366940 330460 0 18 2 0 10 4.06E-07 1.53E-03 BRAF 1137340 320825 0 13 0 0 4 1.08E-06 3.38E-03 TRAF3 858130 216275 0 9 0 0 3 1.35E-05 3.64E-02 -
[1] Brody H. Multiple myeloma[J]. Nature, 2011, 480(7377):S33. DOI: 10.1038/480S33a
[2] Rajkumar S. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management[J]. Am J Hematol, 2020, 95:548-567. DOI: 10.1002/ajh.25791
[3] Gerecke C, Fuhrmann S, Strifler S, et al. The diagnosis and treatment of multiple myeloma[J]. DtschArztebl Int, 2016, 113(27/28):470-476.
[4] DimopoulosMA, Moreau P, Terpos E, et al. Multiple myeloma: EHA-ESMO Clinical Practice Guidelines for diagnosis, treatment and follow-up[J]. Ann Oncol, 2021, 32(3):309-322. DOI: 10.1016/j.annonc.2020.11.014
[5] Martínez-Jiménez F, Muiños F, Sentís I, et al. A compendium of mutational cancer driver genes[J]. Nat Rev Cancer, 2020, 20(10):555-572. DOI: 10.1038/s41568-020-0290-x
[6] PonJR, MarraMA. Driver and passenger mutations in cancer[J]. Annu Rev Pathol, 2015, 10:25-50. DOI: 10.1146/annurev-pathol-012414-040312
[7] Abdallah N, Rajkumar SV, Greipp P, et al. Cytogenetic abnormalities in multiple myeloma: association with disease characteristics and treatment response[J]. Blood Cancer J, 2020, 10(8):82. DOI: 10.1038/s41408-020-00348-5
[8] Höllein A, TwardziokSO, Walter W, et al. The combination of WGS and RNA-Seq is superior to conventional diagnostic tests in multiple myeloma: ready for prime time[J]? Cancer Genet, 2020, 242: 15-24.
[9] LohrJG, Stojanov P, Carter SL, et al. Widespread genetic heterogeneity in multiple myeloma: implications for targeted therapy[J]. Cancer Cell, 2014, 25(1):91-101. DOI: 10.1016/j.ccr.2013.12.015
[10] Tamborero D, Gonzalez-Perez A, Lopez-Bigas N. OncodriveCLUST: exploiting the positional clustering of somatic mutations to identify cancer genes[J]. Bioinformatics, 2013, 29(18):2238-2244. DOI: 10.1093/bioinformatics/btt395
[11] van den Eynden J, Fierro AC, Verbeke LP, et al. SomInaClust: detection of cancer genes based on somatic mutation patterns of inactivation and clustering[J]. BMC Bioinformatics, 2015, 16:125.
[12] LawrenceMS, Stojanov P, Polak P, et al. Mutational heterogeneity in cancer and the search for new cancer-associated genes[J]. Nature, 2013, 499(7457):214-218. DOI: 10.1038/nature12213
[13] Mayakonda A, Lin DC, Assenov Y, et al. Maftools: efficient and comprehensive analysis of somatic variants in cancer[J]. Genome Res, 2018, 28(11):1747-1756. DOI: 10.1101/gr.239244.118
[14] WalkerBA, Mavrommatis K, Wardell CP, et al. Identification of novel mutational drivers reveals oncogene dependencies in multiple myeloma[J]. Blood, 2018, 132(13):1461.
[15] Soussi T, WimanKG. TP53: an oncogene in disguise[J]. Cell Death Differ, 2015, 22(8):1239-1249. DOI: 10.1038/cdd.2015.53
[16] Zhou YJ, Yang ZW, Zhang HL, et al. DNMT3A facilitates colorectal cancer progression via regulating DAB2IP mediated MEK/ERK activation[J]. BiochimBiophys Acta Mol Basis Dis, 2022, 1868(4):166353. DOI: 10.1016/j.bbadis.2022.166353
[17] LeyTJ, Ding L, Walter MJ, et al. DNMT3A mutations in acute myeloid leukemia[J]. N Engl J Med, 2010, 363(25):2424-2433. DOI: 10.1056/NEJMoa1005143
[18] Belizaire R, KoochakiSHJ, UdeshiND, et al. CBL mutations drive PI3K/AKT signaling via increased interaction with LYN and PIK3R1[J]. Blood, 2021, 137(16):2209-2220. DOI: 10.1182/blood.2020006528
[19] KangJH, Lee SH, Lee J, et al. The mutation of BCOR is highly recurrent and oncogenic in mature T-cell lymphoma[J]. BMC Cancer, 2021, 21(1):82. DOI: 10.1186/s12885-021-07806-8
[20] NeuseCJ, Lomas OC, Schliemann C, et al. Genome instability in multiple myeloma[J]. Leukemia, 2020, 34(11):2887-2897. DOI: 10.1038/s41375-020-0921-y
[21] Manier S, Salem KZ, Park J, et al. Genomic complexity of multiple myeloma and its clinical implications[J]. Nat Rev Clin Oncol, 2017, 14(2):100-113. DOI: 10.1038/nrclinonc.2016.122



 下载:
下载: